Definition von Mitochondrien-DNA
Verschiedenes / / July 04, 2022
Begriffsdefinition
Das mitochondriale Genom ist ein kleines, geschlossenes, kreisförmiges Doppelhelix-Molekül mit 16.569 Basenpaaren (bp), das nur von Müttern an Söhne und Töchter (selten) vererbt wird. Fälle von Heteroplasmie), so dass es zum Zeitpunkt der Empfängnis keine genetische Rekombination gibt, sie entwickelt sich auch ausschließlich durch Akkumulation von Mutationen in der Wetter.






Lic. in der Physikalischen Anthropologie
Die hohe Mutationsrate des mitochondrialen Genoms (10- bis 20-mal so hoch wie die der DNS nukleär, in Bezug auf Gene mit vergleichbaren Funktionen) ist nützlich, um zwischen Populationen zu unterscheiden, die im Laufe der Zeit auftreten sind biologisch verwandt, ihre Mutationsrate kann jedoch so schnell sein, dass Mutationsphänomene auftreten können. Rückmutation. Die Rate der Evolution Der Mittelwert dieses Moleküls wurde aus Arten berechnet, bei denen Divergenzzeiten von fossilen Überresten verfügbar waren, und aus biodemografischen oder Proteindaten; das Ergebnis ergibt 1-2% pro Million Jahre, gültig für verschiedene Ordnungen.
Diese bestimmte DNA fungiert als Werkzeug dazu Forschung die biologische Verwandtschaft zwischen Populationen, weil ihre Eigenschaften es ermöglichen, die Beziehungen zwischen Populationen aufzuklären die in letzter Zeit auseinandergegangen sind, ohne Rekombinationsphänomene berücksichtigen und eine Dimension hinzufügen zu müssen vorübergehend; Dazu muss Folgendes beachtet werden: Zwei Personen, deren gemeinsamer Vorfahre eine Frau war, werden haben Mitochondrien-DNA-Moleküle so unterschiedlich wie die Zeit seit der Trennung der Vorfahr.
Haplogruppen für die mitochondriale DNA-Klassifizierung in geografischer Verteilung
Einige DNA-Regionen können einander sehr ähnlich sein, was ihre Einstufung im selben Cluster, der als Haplogruppe bekannt ist. Zum Beispiel haben Torroni et al. (1993) die mitochondrialen Gründungslinien für den amerikanischen Kontinent typisiert, die die die Bezeichnung der Haplogruppen A, B, C und D erhielten, gemäß Punktmutationen der Sequenz mitochondrial. Diese Mutationen schaffen unterschiedliche Spaltungsstellen für spezifische Enzyme, wie unten beschrieben: In Linie A gibt es die Stelle Cutoff 663 für das Enzym HaeIII, Linie C ist durch die Stelle 13259 für HincII gekennzeichnet, in Linie D wird Stelle 5176 erkannt AluI; diese Identifizierung erfolgt anhand von Fragment-Length-Polymorphismen (RFLP). Im Fall von Linie B gibt es eine Deletion von 9 Basenpaaren an Position 8272-8289.
Die geografische Verteilung jeder Linie wurde wie folgt beschrieben: Linie A ist auf dem amerikanischen Kontinent und insbesondere in Nordamerika vorherrschend; Diese Haplogruppe wird jedoch auch mesoamerikanischen Populationen zugeschrieben. Die Linien C und D kommen hauptsächlich in Südamerika vor; Linie B kommt in der nördlichen und südlichen Region der Pazifikküste vor, und Kemp et al. (2010) schlagen sie als charakteristische Linie der vor Familie Yuto-Azteken und Südwesten der USA
Darüber hinaus gibt es Mutationen, die von Individuen derselben Haplogruppe geteilt werden, wodurch es möglich wurde, bestimmte Haplotypen (oder Unterlinien) einiger Populationen zu beschreiben. Abbildung 1 zeigt die Regionen der mitochondrialen DNA, die die Mutationen enthalten, um jede Linie zu erkennen.
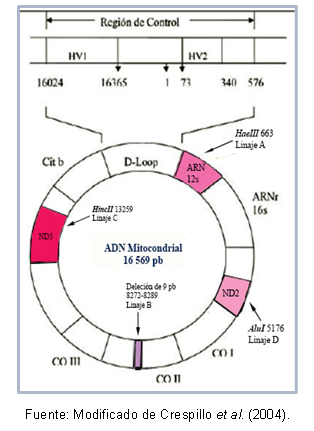
Abbildung 1. Mitochondriale DNA-Regionen, die für jede amerikanische Gründungslinie typisch sind.
Einige Jahre lang wurden nur diese vier Linien anerkannt, bis die Haplogruppe X für die nördlichen Populationen des Kontinents aufgenommen wurde; die auch entfernt mit europäischen Populationen verwandt ist. Die vorherigen Linien haben eine Entsprechung mit asiatischen Populationen, obwohl sie weniger häufig beobachtet werden als auf dem amerikanischen Kontinent; die Abstammungslinien A, B und C werden bei modernen Afrikanern und Kaukasiern nicht gefunden; und die Linie D existiert auch in Afrika, ist aber mit anderen Restriktionsstellen assoziiert. Mit dem oben Gesagten könnten wir sagen, dass diese Haplogruppen die amerikanische Bevölkerung charakterisieren und daher ihre Untersuchung in Bezug auf Besiedlung und Migration (alte und neuere) angemessen ist.
genetische Distanzen
Der Weg, um genetische Ähnlichkeiten oder Unterschiede zwischen Populationen festzustellen, führt über genetische Distanzen, die a historische Erklärung, da sie sich im Laufe der Generationen ändern (zunehmen oder verringern) und uns zu Ereignissen in der Geschichte führen können von einem Population zum beispiel: bei einer großen migration oder einem kontakt zwischen zwei kulturen können wir mit dieser rechnung erkennen, welche mechanismen in aktion getreten sind, um uns dieses oder jenes ergebnis zu geben.
Sowohl Gendrift als auch Genfluss haben mit der Leichtigkeit zu tun, mit der sich eine Gruppe, in diesem Fall Menschen, innerhalb einer Gruppe bewegen muss. Gebiet und mit anderen Gruppen in Kontakt zu treten. Geografische Isolation ist also die genetische Distanz, die mit zunehmender geografischer Distanz zwischen menschlichen Gruppen wächst.
Mitochondriale DNA-Diversitätshypothese
Zur Erklärung der Diversität in den Gründungslinien der Mitochondrien in Amerika gibt es zwei Hypothese: dass dieser Kontinent durch mehrere Ereignisse von Beringia kolonisiert wurde oder dass nach der Migration evolutionäre Veränderungen nach der Kolonisierung aufgetreten sind. Zwei Eintrittswege dieser Variationen auf den Kontinent werden ebenfalls erklärt, die erste schlägt vor, dass die vier Gründungs-Haplogruppen ohne Variationen, d. h. jede mit einer Root-Haplotyp, sie könnten kurz nach dem Maximum der letzten Eiszeit oder kurz davor angekommen sein, mit Daten von vor 21.000 bis 19.000 Jahren und wären einer Küstenroute durch die gefolgt Friedlich; Der zweite Vorschlag legt nahe, dass diese Variationen innerhalb der Haplogruppe bereits in Beringia existierten und in den Süden des amerikanischen Kontinents gebracht wurden, aber ihr Eintritt wäre gewesen genau am Ende des letzten Gletschermaximums, so dass die Routen innerhalb des Kontinents bereits frei wären, dann wäre der Eintritt dieser Menschengruppen vor 19.000 Jahre. Es gibt auch eine große Vielfalt der Haplogruppe A und eine Koaleszenzzeit, die kürzer ist als die der anderen (17.000 Jahre).
Die wahrscheinlichste Erklärung dafür ist, dass dies auf sekundäre Expansionen der Haplogruppe A von Beringia lange nach dem Ende des letzten Gletschermaximums zurückzuführen ist. Trotz der Diskrepanz in Bezug auf den Zeitpunkt der Einreise des Menschen in Amerika haben genetische Studien etwas Klarheit, weil sie die Hypothese stützen, dass es schon früher menschliche Gruppen auf dem amerikanischen Kontinent gab Clovis; und es wurde festgestellt, dass es eine Trennung zwischen den Vorfahren Nordostasiens vor 25-35.000 Jahren und dem Eintritt in Amerika vor 15.000-35.000 Jahren gibt. Abbildung 2 zeigt die Routen der mitochondrialen Haplogruppen in der Welt und den Zeitpunkt der Divergenz in Jahren vor der Gegenwart.
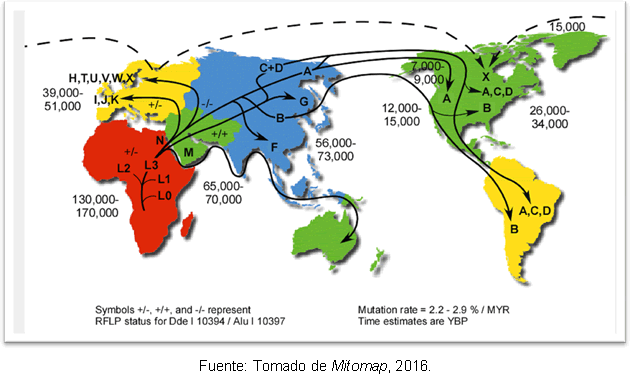
Figur 2. Karte der verschiedenen Verbreitungswege der mitochondrialen Abstammungslinien
Bedeutung der Phylogeographie in genetischen Studien und mitochondrialen Gendaten
Es gibt ein sehr nützliches Werkzeug für die genetische Analyse; die Phylogeographie. Dies sind die ersten Anwendungen molekularer Studien, und damit versuchen wir, sie bestimmen zu können die phylogenetischen und räumlichen Beziehungen zwischen Nukleotidsequenzen, in diesem Fall DNA mitochondrial. Die räumliche Verteilung kann einem zeitlichen Muster ähneln, das heißt, die geographisch am weitesten entfernten DNA-Sequenzen sollten am weitesten entfernt sein genetisch unterschiedlich, sowie DNA-Sequenzen, die vor langer Zeit auseinandergegangen sind, sollten auch am unterschiedlichsten sein genetisch. So würden geografisch entfernte Populationen mit wenig oder keinem Genfluss zwischen ihnen Unterschiede aufgrund von genetischer Drift und Mutation akkumulieren, selbst durch Selektion; Es können jedoch Situationen auftreten, die keine Drift zulassen, wie z. B. ein oder mehrere Gründereffekte oder andere Muster des Genflusses.
Die von mitochondrialen Genen bereitgestellten Daten gehören aufgrund der Charakteristisch für diese Gene ist, dass sie sich nicht rekombinieren und daher eine viel klarere phylogenetische Linie aufweisen als viele andere Kerngene. Die effektive Populationsgröße, die mit den mitochondrialen Genen berechnet wird (und mit dem Y-Chromosom auf ähnliche Weise funktioniert), beträgt etwa ein Viertel derjenigen, die für die mitochondrialen Gene berechnet wurde. Kerngene, dann tritt die Divergenz fast viermal schneller auf als bei Kerngenen, die diese schnelle Rate der Divergenz (und des Genflusses) verursachen können Das beobachtete Vererbungsmuster in diesen uniparentalen Genen unterscheidet sich von den Phylogenien, die mit Kerngenen erhalten werden (die den Großteil des Genpools in a Individuell).
Verweise
Achilli, A., Perego, U. A., Bravi, C. M., Kobel, M. D., Kong, Q.-P., & et al. (2008). Die Phylogenie der vier panamerikanischen MtDNA-Haplogruppen: Implikationen für Evolutions- und Krankheitsstudien. PLoS ONE, 3(3), e1764.Cavalli-Sforza, L. L. (2000). Gene, Völker und Sprachen (1. Auflage in Pocket Library ed.). Barcelona, Spanien: Kritischer Leitartikel.
Rebato, E., Susanne, C., & Chiarelli, B. (Bearbeitungen.). (2005). Biologische Anthropologie verstehen: Evolution und Humanbiologie. Göttliches Wort.
Torroni, A., Schürr, T. G., Cabell, M. F, Braun, M. D, Nel, J. V., Larsen, M., et al. (1993). Asiatische Affinitäten und kontinentale Ausstrahlung der vier Gründungs-MtDNAs der amerikanischen Ureinwohner. The American Journal of Human Genetics, 53(3), 563.
(2022). mitochondriale DNA, Nationales Institut für Humangenomforschung.
-Mitochondriale DNA schematische Referenz-
Crespillo, M., Paredes, M., Arimany, J., Guerrero, L., & Valverde, J. (2004). Spanischer Bürgerkrieg (1936-1939): Identifizierung menschlicher Überreste aus Massengräbern in Katalonien durch mitochondriale DNA-Analyse. Über einen Fall. Notizbücher für Gerichtsmedizin (38), 37-46.
