Definizione di DNA mitocondriale
Varie / / July 04, 2022
definizione del concetto
Il genoma mitocondriale è una piccola molecola circolare chiusa a doppia elica di 16.569 paia di basi (bp) che viene ereditata solo dalle madri ai figli e alle figlie (raramente). casi di eteroplasmia), quindi non c'è ricombinazione genetica al momento del concepimento, inoltre si evolve esclusivamente per accumulo di mutazioni nel tempo atmosferico.






Lic. in Antropologia Fisica
L'alto tasso di mutazione del genoma mitocondriale (da 10 a 20 volte quello del DNA nucleare, in termini di geni con funzioni comparabili) è utile per differenziare tra popolazioni che nel tempo sono stati biologicamente correlati, tuttavia, il loro tasso di mutazione può essere così veloce che possono verificarsi fenomeni di mutazione. retromutazione. Il tasso di Evoluzione La media di questa molecola è stata calcolata da specie in cui erano disponibili tempi di divergenza dai resti fossili e da dati biodemografici o proteici; il risultato dà 1-2% per milione di anni, valido per ordini diversi.
Questo particolare DNA funziona come uno strumento per ricerca la parentela biologica tra le popolazioni perché le loro proprietà consentono di chiarire le relazioni tra le popolazioni divergenti negli ultimi tempi, senza dover considerare fenomeni di ricombinazione e aggiungere una dimensione temporaneo; Per questo, è necessario tenere presente quanto segue: avranno due individui il cui antenato comune è stata una donna molecole di DNA mitocondriale diverse quanto il tempo è trascorso dalla separazione del antenato.
Aplogruppi per la classificazione del DNA mitocondriale nella distribuzione geografica
Alcune regioni del DNA possono essere molto simili tra loro, consentendo loro classificazione nello stesso cluster, noto come aplogruppo. Ad esempio, Torroni et al (1993) hanno caratterizzato i lignaggi mitocondriali fondatori per il continente americano, il che ha ricevuto la denominazione di aplogruppi A, B, C e D, in base alle mutazioni puntiformi della sequenza mitocondriale. Queste mutazioni creano diversi siti di scissione per enzimi specifici, come descritto di seguito: nel lignaggio A c'è il sito cutoff 663 per l'enzima HaeIII, il lignaggio C è caratterizzato dal sito 13259 per HincII, nel lignaggio D il sito 5176 è riconosciuto per AluI; questa identificazione è fatta da polimorfismi di lunghezza del frammento (RFLP). Nel caso del lignaggio B, c'è una cancellazione di 9 coppie di basi nella posizione 8272-8289.
La distribuzione geografica di ciascun ceppo è stata così descritta: il ceppo A è predominante nel Continente Americano e soprattutto nel Nord America; tuttavia, questo aplogruppo è attribuito anche alle popolazioni mesoamericane. I lignaggi C e D compaiono principalmente in Sud America; il lignaggio B si trova nella regione settentrionale e meridionale della costa del Pacifico e Kemp et al (2010) lo propone come un lignaggio caratteristico del famiglia Yuto-azteco e Stati Uniti sudoccidentali
Inoltre, esistono mutazioni condivise tra individui dello stesso aplogruppo, il che ha permesso di descrivere aplotipi (o sottolinee) specifici di alcune popolazioni. La Figura 1 mostra le regioni del DNA mitocondriale che contengono le mutazioni per riconoscere ogni lignaggio.
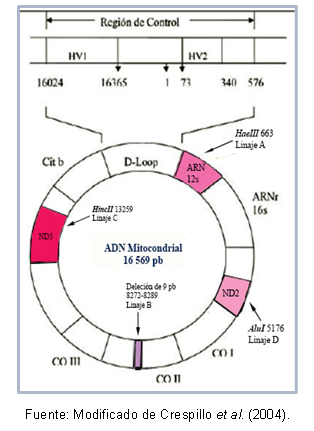
Figura 1. Regioni del DNA mitocondriale che caratterizzano ogni lignaggio fondatore americano.
Per alcuni anni furono riconosciuti solo questi quattro lignaggi fino all'inclusione dell'aplogruppo X per le popolazioni settentrionali del continente; che è anche lontanamente imparentato con le popolazioni europee. I ceppi precedenti hanno una corrispondenza con popolazioni asiatiche, sebbene siano osservati meno frequentemente che nel Continente americano; i lignaggi A, B e C non si trovano negli africani e nei caucasici moderni; e il lignaggio D esiste anche in Africa, ma è associato ad altri siti di restrizione. Con quanto sopra, potremmo dire che questi aplogruppi caratterizzano le popolazioni americane e quindi il loro studio è adeguato in termini di insediamenti e migrazioni (vecchie e recenti).
distanze genetiche
Il modo per stabilire somiglianze o differenze genetiche tra le popolazioni è con le distanze genetiche, che possono avere a spiegazione storica, poiché cambiano (crescono o diminuiscono) nel corso delle generazioni e possono condurci a eventi della storia di uno popolazione per esempio: nel momento di una grande migrazione o di contatto tra due culture, con questo calcolo possiamo discernere quali meccanismi sono entrati in azione per darci questo o quel risultato.
Sia la deriva genetica che il flusso genico hanno a che fare con la facilità con cui un gruppo, in questo caso gli esseri umani, deve muoversi all'interno di un gruppo. territorio ed entrare in contatto con altri gruppi. Quindi, l'isolamento geografico è la distanza genetica che cresce con l'aumentare della distanza geografica tra i gruppi umani.
Ipotesi della diversità del DNA mitocondriale
Per spiegare il diversità nei lignaggi mitocondriali fondatori in America, ce ne sono due ipotesi: che questo continente sia stato colonizzato da molteplici eventi provenienti dalla Beringia, o che una volta avvenuta la migrazione, si siano verificati cambiamenti evolutivi dopo la colonizzazione. Vengono spiegate anche due vie di ingresso di queste variazioni nel continente, la prima propone che i quattro aplogruppi fondatori senza variazioni, cioè ciascuno con un aplotipo radicale, sarebbero potuti arrivare subito dopo l'Ultimo Massimo Glaciale o poco prima, con date comprese tra 21mila e 19mila anni fa e avrebbero seguito una rotta costiera dal Tranquillo, calmo; la seconda proposta suggerisce che queste variazioni intra-aplogruppi esistessero già in Beringia e furono portate nel sud del continente americano ma il loro ingresso sarebbe stato esattamente alla fine dell'Ultimo Massimo Glaciale in modo che le rotte all'interno del continente fossero già libere, quindi l'ingresso di questi gruppi umani sarebbe 19mila fa anni. C'è anche una grande diversità di aplogruppo A e un tempo di coalescenza che è più breve del resto (17 mila anni).
La spiegazione più probabile di ciò è che è dovuto alle espansioni secondarie dell'aplogruppo A dalla Beringia, molto tempo dopo la fine dell'ultimo massimo glaciale. Nonostante la discrepanza riguardo all'ora dell'ingresso dell'uomo nelle Americhe, gli studi genetici lo hanno fatto fanno un po' di chiarezza, perché supportano l'ipotesi che prima esistessero gruppi umani nel continente americano Clodoveo; e si scopre che c'è una separazione tra gli antenati dell'Asia nord-orientale 25-35 mila anni fa e l'ingresso in America 15-35 mila anni fa. La figura 2 mostra le rotte degli aplogruppi mitocondriali nel mondo e il tempo di divergenza negli anni prima del presente.
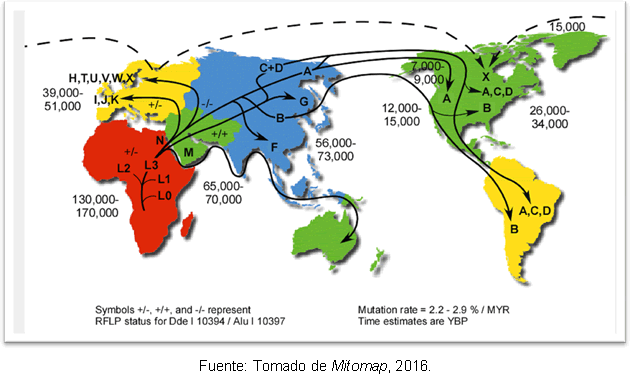
Figura 2. Mappa delle diverse vie di dispersione dei lignaggi mitocondriali
Importanza della filogeografia nello studio genetico e nei dati sui geni mitocondriali
Esiste uno strumento molto utile per l'analisi genetica; la filogeografia. Queste sono le prime applicazioni degli studi molecolari e con essi cerchiamo di essere in grado di determinare le relazioni filogenetiche e spaziali tra le sequenze nucleotidiche, in questo caso il DNA mitocondriale. La distribuzione spaziale può assomigliare a uno schema temporale, ovvero le sequenze di DNA geograficamente più distanti dovrebbero essere le più distanti geneticamente diverse, così come le sequenze di DNA divergenti molto tempo fa dovrebbero anche essere le più diverse geneticamente. Quindi popolazioni geograficamente distanti, con poco o nessun flusso genico tra di loro, accumulerebbero differenze dovute alla deriva genetica e alla mutazione, anche per selezione; ma possono verificarsi situazioni che non consentono la deriva, come uno o più effetti fondatore o altri modelli di flusso genico.
I dati forniti dai geni mitocondriali sono stati tra i più utili nella ricerca filogeografia a causa del caratteristica che questi geni hanno di non ricombinarsi e di conseguenza mostrano una linea filogenetica molto più chiara di molti geni nucleari. La dimensione effettiva della popolazione calcolata con i geni mitocondriali (e con il cromosoma Y che funziona in modo simile) è circa un quarto di quella calcolata per i geni mitocondriali. geni nucleari, quindi la divergenza si verifica quasi quattro volte più velocemente rispetto ai geni nucleari, questo rapido tasso di divergenza (e flusso genico) può causare il il modello di ereditarietà osservato in questi geni uniparentali è diverso dalle filogenesi ottenute con i geni nucleari (che rappresentano la maggior parte del pool genico in un individuale).
Riferimenti
Achille, A., Perego, U. A., Bravi, C. M., Coble, M. D., Kong, Q.-P., & et al. (2008). La filogenesi dei quattro aplogruppi panamericani dell'MtDNA: implicazioni per gli studi evolutivi e sulle malattie. PLoS UNO, 3(3), e1764.Cavalli-Sforza, L. l. (2000). Geni, popoli e linguaggi (1a Edizione in Pocket Library ed.). Barcellona, Spagna: Editoriale critico.
Rebato, E., Susanne, C., & Chiarelli, B. (Modifiche.). (2005). Per comprendere l'antropologia biologica: evoluzione e biologia umana. Parola divina.
Torroni, A., Schurr, T. G., Cabell, M. F, Marrone, M. D, Neel, J. V., Larsen, M., et al. (1993). Affinità asiatiche e radiazione continentale dei quattro mtDNA nativi americani fondatori. The American Journal of Human Genetics, 53(3), 563.
(2022). DNA mitocondriale, Istituto nazionale di ricerca sul genoma umano.
-Riferimento schematico del DNA mitocondriale-
Crespillo, M., Paredes, M., Arimany, J., Guerrero, L., & Valverde, J. (2004). Guerra civile spagnola (1936-1939): identificazione di resti umani da fosse comuni in Catalogna mediante analisi del DNA mitocondriale. A proposito di un caso. Quaderni di medicina legale (38), 37-46.



