Definitie van mitochondriaal DNA
Diversen / / July 04, 2022
conceptdefinitie
Het mitochondriale genoom is een klein, gesloten, cirkelvormig molecuul met dubbele helix van 16.569 basenparen (bp) dat alleen wordt geërfd van moeders op zonen en dochters (zelden). gevallen van heteroplasmie), dus er is geen genetische recombinatie op het moment van conceptie, het evolueert ook uitsluitend door accumulatie van mutaties in de het weer.






lic. in fysieke antropologie
De hoge mate van mutatie van het mitochondriale genoom (10 tot 20 keer die van de DNA nucleair, in termen van genen met vergelijkbare functies) is nuttig om onderscheid te maken tussen populaties die in de loop van de tijd biologisch verwant zijn, maar hun mutatiesnelheid kan zo snel zijn dat mutatieverschijnselen kunnen optreden. terugmutatie. het tarief van evolutie Het gemiddelde van dit molecuul is berekend uit soorten waarin afwijkende tijden van fossiele resten beschikbaar waren, en uit biodemografische of eiwitgegevens; het resultaat geeft 1-2% per miljoen jaar, geldig voor verschillende bestellingen.
Dit specifieke DNA functioneert als een hulpmiddel om Onderzoek de biologische verwantschap tussen populaties omdat hun eigenschappen het mogelijk maken de relaties tussen populaties op te helderen die de afgelopen tijd zijn uiteengelopen, zonder rekening te hoeven houden met recombinatieverschijnselen en een dimensie toe te voegen tijdelijk; Hiervoor moet het volgende in gedachten worden gehouden: twee individuen van wie de gemeenschappelijke voorouder een vrouw is, zullen hebben: mitochondriale DNA-moleculen zo verschillend als de tijd is verstreken sinds de scheiding van de Voorouder.
Haplogroepen voor mitochondriaal DNA-classificatie in geografische spreiding
Sommige DNA-gebieden kunnen erg op elkaar lijken, waardoor hun classificatie in dezelfde cluster, die bekend staat als een haplogroep. Torroni et al (1993) typeerden bijvoorbeeld de oprichtende mitochondriale lijnen voor het Amerikaanse continent, de die de benaming van haplogroepen A, B, C en D ontving, volgens puntmutaties van de sequentie mitochondriaal. Deze mutaties creëren verschillende splitsingsplaatsen voor specifieke enzymen, zoals hieronder beschreven: in lijn A is er de plaats grenswaarde 663 voor het enzym HaeIII, lijn C wordt gekenmerkt door plaats 13259 voor HincII, in lijn D wordt plaats 5176 herkend voor AluI; deze identificatie is gemaakt van fragmentlengtepolymorfismen (RFLP). In het geval van afstamming B is er een deletie van 9 basenparen op positie 8272-8289.
De geografische spreiding van elke afstamming is als volgt beschreven: afstamming A is overheersend op het Amerikaanse continent en vooral in Noord-Amerika; deze haplogroep wordt echter ook toegeschreven aan Meso-Amerikaanse populaties. Lineages C en D komen voornamelijk voor in Zuid-Amerika; afstamming B wordt gevonden in de noordelijke en zuidelijke regio van de Pacifische kust, en Kemp et al (2010) stellen het voor als een karakteristieke afstamming van de familie Yuto-Aztecan en het zuidwesten van de V.S.
Daarnaast zijn er mutaties die worden gedeeld tussen individuen van dezelfde haplogroep, wat het mogelijk heeft gemaakt om specifieke haplotypes (of sublijnen) van sommige populaties te beschrijven. Figuur 1 toont de regio's van het mitochondriale DNA die de mutaties bevatten om elke afstamming te herkennen.
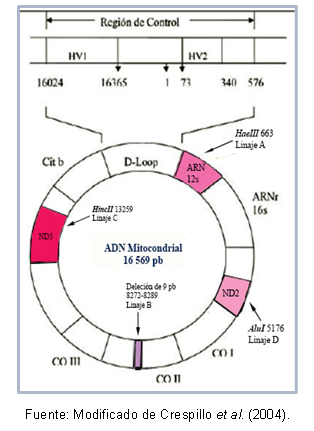
Figuur 1. Mitochondriale DNA-regio's die elke Amerikaanse stamvader typeren.
Gedurende enkele jaren werden alleen deze vier geslachten erkend totdat haplogroep X werd opgenomen voor de noordelijke populaties van het continent; die ook in de verte verwant is aan de Europese bevolking. De vorige geslachten hebben een overeenkomst met Aziatische populaties, hoewel ze minder vaak worden waargenomen dan op het Amerikaanse continent; geslachten A, B en C worden niet gevonden in moderne Afrikanen en blanken; en afstamming D bestaat ook in Afrika, maar is geassocieerd met andere restrictieplaatsen. Met het bovenstaande zouden we kunnen zeggen dat deze haplogroepen kenmerkend zijn voor de Amerikaanse populaties en daarom is hun studie adequaat in termen van vestiging en migraties (oud en recent).
genetische afstanden
De manier om genetische overeenkomsten of verschillen tussen populaties vast te stellen, is met genetische afstanden, die een historische verklaring, omdat ze in de loop van de generaties veranderen (toenemen of afnemen) en ons naar gebeurtenissen in de geschiedenis kunnen leiden van een bevolking bijvoorbeeld: op het moment van een grote migratie of contact tussen twee culturen, kunnen we met deze berekening onderscheiden welke mechanismen in actie zijn gekomen om ons dit of dat resultaat te geven.
Zowel genetische drift als gene flow hebben te maken met het gemak waarmee een groep, in dit geval mensen, zich binnen een groep moet verplaatsen. grondgebied en om in contact te komen met andere groepen. Geografische isolatie is dus de genetische afstand die groeit met toenemende geografische afstand tussen menselijke groepen.
Mitochondriaal DNA-diversiteitshypothese
om het uit te leggen diversiteit in de oprichtende mitochondriale lijnen in Amerika zijn er twee: hypothese: dat dit continent is gekoloniseerd door meerdere gebeurtenissen uit Beringia, of dat zodra migratie heeft plaatsgevonden, er evolutionaire veranderingen zijn opgetreden na kolonisatie. Twee routes van binnenkomst van deze variaties op het continent worden ook uitgelegd, de eerste stelt voor dat de vier oprichtende haplogroepen zonder variaties, dat wil zeggen, elk met een wortelhaplotype, kunnen ze net na het Laatste Glaciale Maximum zijn aangekomen of iets eerder, met data van 21 duizend tot 19 duizend jaar geleden, en zouden ze een kustroute hebben gevolgd door de Vredevol; het tweede voorstel suggereert dat deze variaties binnen de haplogroep al bestonden in Beringia en naar het zuiden van het Amerikaanse continent werden gebracht, maar dat hun intrede zou zijn geweest precies aan het einde van het Laatste Glaciale Maximum zodat de routes binnen het continent al vrij zouden zijn, dan zou de intrede van deze menselijke groepen 19 duizend geleden zijn jaar. Er is ook een grote diversiteit aan haplogroep A en een samensmeltingstijd ervoor die korter is dan de rest (17 duizend jaar).
De meest waarschijnlijke verklaring hiervoor is dat het te wijten is aan secundaire uitbreidingen van haplogroep A uit Beringia, lang na het einde van het Laatste Glaciale Maximum. Ondanks de discrepantie met betrekking tot het tijdstip waarop de mens Amerika binnenkwam, hebben genetische studies enige duidelijkheid geven, omdat ze de hypothese ondersteunen dat er eerder menselijke groepen op het Amerikaanse continent waren Clovis; en het is gebleken dat er een scheiding is tussen de voorouders van Noordoost-Azië 25-35 duizend jaar geleden en de intrede in Amerika 15-35 duizend jaar geleden. Figuur 2 toont de routes van de mitochondriale haplogroepen in de wereld en de tijd van divergentie in jaren vóór het heden.
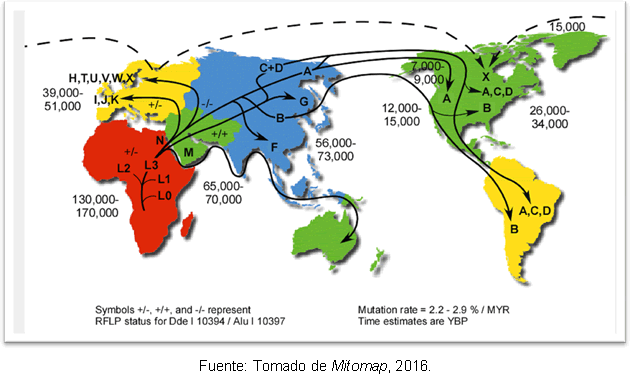
Figuur 2. Kaart van de verschillende routes van verspreiding van mitochondriale lijnen
Belang van fylogeografie in genetische studie en mitochondriale gengegevens
Er is een zeer nuttig hulpmiddel voor genetische analyse; de fylogeografie. Dit zijn de eerste toepassingen van moleculaire studies, en daarmee willen we kunnen bepalen: de fylogenetische en ruimtelijke relaties tussen nucleotidesequenties, in dit geval DNA mitochondriaal. De ruimtelijke verdeling kan lijken op een temporeel patroon, dat wil zeggen dat de meest geografisch verwijderde DNA-sequenties het meest moeten zijn genetisch verschillend, evenals DNA-sequenties die lang geleden uiteenliepen, zouden ook het meest verschillend moeten zijn genetisch. Dus geografisch ver verwijderde populaties, met weinig of geen genenstroom tussen hen, zouden verschillen accumuleren als gevolg van genetische drift en mutatie, zelfs door selectie; maar er kunnen zich situaties voordoen die drift niet toestaan, zoals een of meer grondleggereffecten of andere patronen van gene flow.
De gegevens die door mitochondriale genen worden geleverd, behoren tot de meest bruikbare in fylogeografie-onderzoek vanwege de kenmerk dat deze genen hebben dat ze niet recombineren en als gevolg daarvan vertonen ze een veel duidelijkere fylogenetische lijn dan veel nucleaire genen. De effectieve populatiegrootte berekend met de mitochondriale genen (en met het Y-chromosoom dat op een vergelijkbare manier functioneert) is ongeveer een kwart van die berekend voor de mitochondriale genen. nucleaire genen, dan vindt divergentie bijna vier keer sneller plaats dan bij nucleaire genen, kan deze snelle mate van divergentie (en genstroom) de waargenomen patroon van overerving in deze uniparentale genen is anders dan de fylogenieën die worden verkregen met nucleaire genen (die het grootste deel van de genenpool in een individueel).
Referenties
Achilli, A., Perego, U. A., Bravi, C. M., Coble, M. D., Kong, Q.-P., & et al. (2008). De fylogenie van de vier pan-Amerikaanse MtDNA-haplogroepen: implicaties voor evolutionaire en ziektestudies. PLoS EEN, 3(3), e1764.Cavalli-Sforza, L. L. (2000). Genen, volkeren en talen (1e editie in Pocket Library red.). Barcelona, Spanje: kritische redactie.
Rebato, E., Susanne, C., & Chiarelli, B. (Bewerkingen.). (2005). Biologische antropologie begrijpen: evolutie en menselijke biologie. Goddelijk Woord.
Torroni, A., Schurr, T. G., Cabell, M. F, Bruin, M. D, Neel, J. V., Larsen, M., et al. (1993). Aziatische affiniteiten en continentale straling van de vier oorspronkelijke Amerikaanse mtDNA's. The American Journal of Human Genetics, 53 (3), 563.
(2022). mitochondriaal DNA, National Human Genome Research Institute.
-Mitochondriaal DNA schematische referentie-
Crespillo, M., Paredes, M., Arimany, J., Guerrero, L., & Valverde, J. (2004). Spaanse Burgeroorlog (1936-1939): identificatie van menselijke resten uit massagraven in Catalonië door mitochondriale DNA-analyse. Over een zaak. Notitieboekjes forensische geneeskunde (38), 37-46.

