Definicja mitochondrialnego DNA
Różne / / July 04, 2022
definicja koncepcji
Genom mitochondrialny jest małą, zamkniętą, okrągłą cząsteczką o podwójnej helisie i 16 569 parach zasad (pz), która jest dziedziczona wyłącznie od matek po synów i córki (rzadko). przypadków heteroplazmii), a więc nie ma rekombinacji genetycznej w momencie poczęcia, ewoluuje również wyłącznie przez akumulację mutacji w pogoda.






Lic. w antropologii fizycznej
Wysoki wskaźnik mutacji genomu mitochondrialnego (10 do 20 razy większy niż DNA jądrowe, pod względem genów o porównywalnych funkcjach) jest przydatne do różnicowania populacji, które w czasie są biologicznie spokrewnione, jednak ich tempo mutacji może być tak szybkie, że mogą wystąpić zjawiska mutacji. mutacja wsteczna. Stawka ewolucja Średnia tej cząsteczki została obliczona na podstawie gatunków, dla których dostępne były czasy rozejścia się od szczątków kopalnych, oraz na podstawie danych biodemograficznych lub białkowych; wynik daje 1-2% na milion lat, ważne dla różnych zamówień.
To szczególne DNA działa jako narzędzie do: Badania biologiczne pokrewieństwo między populacjami, ponieważ ich właściwości pozwalają na wyjaśnienie związków między populacjami które rozeszły się w ostatnim czasie, bez konieczności rozważania zjawisk rekombinacji i dodawania wymiaru tymczasowy; W tym celu należy pamiętać o następujących kwestiach: dwie osoby, których wspólnym przodkiem była kobieta, będą miały cząsteczki mitochondrialnego DNA tak różne, jak czas upłynął od oddzielenia przodek.
Haplogrupy do klasyfikacji mitochondrialnego DNA w rozmieszczeniu geograficznym
Niektóre regiony DNA mogą być do siebie bardzo podobne, co pozwala na ich Klasyfikacja w tej samej gromadzie, która jest znana jako haplogrupa. Na przykład Torroni i wsp. (1993) scharakteryzowali założycielskie linie mitochondrialne na kontynencie amerykańskim, które otrzymały nazwę haplogrupy A, B, C i D, zgodnie z punktowymi mutacjami sekwencji mitochondrialny. Mutacje te tworzą różne miejsca cięcia dla określonych enzymów, jak opisano poniżej: w linii A jest miejsce punkt odcięcia 663 dla enzymu HaeIII, linia C charakteryzuje się miejscem 13259 dla HincII, w linii D miejsce 5176 jest rozpoznawane przez Alu; ta identyfikacja jest dokonywana na podstawie polimorfizmów długości fragmentów (RFLP). W przypadku linii B nastąpiła delecja 9 par zasad w pozycji 8272-8289.
Rozkład geograficzny każdej linii został opisany w następujący sposób: linia A dominuje na kontynencie amerykańskim, a zwłaszcza w Ameryce Północnej; jednak ta haplogrupa jest również przypisywana populacjom mezoamerykańskim. Linie C i D występują głównie w Ameryce Południowej; linia B występuje w północnym i południowym regionie wybrzeża Pacyfiku, a Kemp i in. (2010) proponuje ją jako charakterystyczny rodowód rodzina Yuto-Aztecan i południowo-zachodnie Stany Zjednoczone
Ponadto istnieją mutacje, które są wspólne między osobnikami tej samej haplogrupy, co umożliwiło opisanie określonych haplotypów (lub podlinii) niektórych populacji. Rycina 1 pokazuje regiony mitochondrialnego DNA, które zawierają mutacje umożliwiające rozpoznanie każdej linii.
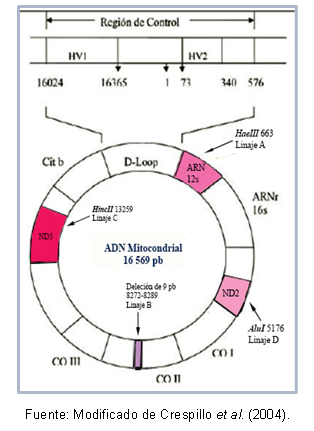
Rysunek 1. Regiony mitochondrialnego DNA charakterystyczne dla każdej amerykańskiej linii założycielskiej.
Przez kilka lat tylko te cztery linie były rozpoznawane, dopóki haplogrupa X nie została włączona do populacji północnej kontynentu; co jest również daleko spokrewnione z populacjami europejskimi. Poprzednie rodowody mają korespondencję z populacjami azjatyckimi, chociaż obserwowane są rzadziej niż na kontynencie amerykańskim; linie A, B i C nie występują u współczesnych Afrykanów i Kaukaskich; i linia D również istnieje w Afryce, ale jest powiązana z innymi miejscami restrykcyjnymi. Z powyższego można by powiedzieć, że te haplogrupy charakteryzują populacje amerykańskie i dlatego ich badanie jest adekwatne pod względem osadnictwa i migracji (starych i niedawnych).
odległości genetyczne
Sposobem na ustalenie podobieństw genetycznych lub różnic między populacjami są odległości genetyczne, które mogą mieć wyjaśnienie historyczne, ponieważ zmieniają się (wzrastają lub maleją) na przestrzeni pokoleń i mogą prowadzić nas do wydarzeń w historii z jednego populacja na przykład: w momencie wielkiej migracji lub kontaktu między dwiema kulturami, dzięki tej kalkulacji możemy rozeznać, jakie mechanizmy zadziałały, aby dać nam taki lub inny wynik.
Zarówno dryf genetyczny, jak i przepływ genów mają związek z łatwością, z jaką grupa, w tym przypadku istoty ludzkie, musi poruszać się w obrębie grupy. terytorium i nawiązać kontakt z innymi grupami. Tak więc izolacja geograficzna to dystans genetyczny, który rośnie wraz ze wzrostem odległości geograficznej między grupami ludzi.
Hipoteza różnorodności mitochondrialnego DNA
Aby wyjaśnić różnorodność w założycielskich liniach mitochondrialnych w Ameryce są dwa hipoteza: że kontynent ten został skolonizowany przez wiele wydarzeń z Beringii, lub że po migracji nastąpiły zmiany ewolucyjne po kolonizacji. Wyjaśnione są również dwie drogi wejścia tych odmian na kontynent, pierwsza sugeruje, że cztery haplogrupy założycielskie bez odmian, to znaczy każda z haplotyp korzeniowy, mogły dotrzeć tuż po ostatnim zlodowaceniu maksimum lub nieco wcześniej, z datami od 21 tys. do 19 tys. lat temu i podążali drogą przybrzeżną przez Spokojna; druga propozycja sugeruje, że te wariacje wewnątrzhaplogrupowe istniały już w Beringii i zostały sprowadzone na południe kontynentu amerykańskiego, ale ich wejście byłoby dokładnie pod koniec ostatniego zlodowacenia, aby trasy w obrębie kontynentu były już wolne, wtedy wejście tych grup ludzkich nastąpiłoby 19 tys. temu lat. Istnieje również duże zróżnicowanie haplogrupy A i krótszy niż pozostałe czas koalescencji (17 tys. lat).
Najbardziej prawdopodobnym wyjaśnieniem tego jest to, że jest to spowodowane wtórną ekspansją haplogrupy A z Beringii, długo po zakończeniu ostatniego zlodowacenia maksimum. Pomimo rozbieżności co do czasu wkroczenia człowieka do obu Ameryk, badania genetyczne dają pewną jasność, ponieważ wspierają hipotezę, że na kontynencie amerykańskim istniały wcześniej grupy ludzkie Chlodwig; i okazuje się, że istnieje separacja między przodkami Azji Północno-Wschodniej 25-35 tysięcy lat temu a wejściem do Ameryki 15-35 tysięcy lat temu. Rycina 2 przedstawia trasy haplogrup mitochondrialnych na świecie oraz czas dywergencji w latach przed teraźniejszością.
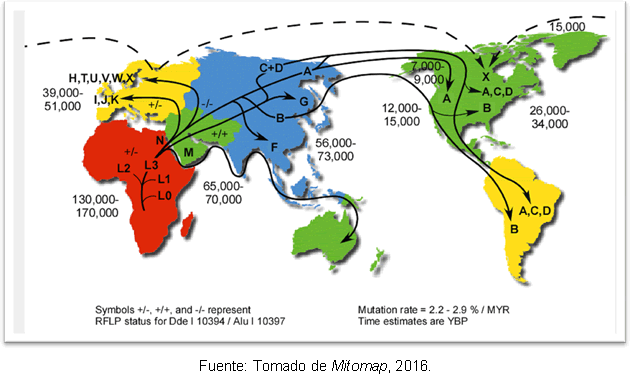
Rysunek 2. Mapa różnych dróg rozprzestrzeniania się linii mitochondrialnych
Znaczenie filogeografii w badaniach genetycznych i mitochondrialnych danych genowych
Istnieje bardzo przydatne narzędzie do analizy genetycznej; filogeografia. Są to pierwsze zastosowania badań molekularnych, a wraz z nimi staramy się być w stanie określić relacje filogenetyczne i przestrzenne między sekwencjami nukleotydów, w tym przypadku DNA mitochondrialny. Rozkład przestrzenny może przypominać wzór czasowy, tzn. najbardziej odległe geograficznie sekwencje DNA powinny być jak najbardziej genetycznie różne, a także sekwencje DNA, które już dawno się rozeszły, również powinny być jak najbardziej odmienne genetycznie. Tak więc populacje odległe geograficznie, z niewielkim lub żadnym przepływem genów między nimi, gromadziłyby różnice z powodu dryfu genetycznego i mutacji, nawet poprzez selekcję; ale mogą wystąpić sytuacje, które nie pozwalają na dryf, takie jak jeden lub więcej efektów założycielskich lub inne wzorce przepływu genów.
Dane dostarczone przez geny mitochondrialne były jednymi z najbardziej przydatnych w badaniach filogeograficznych ze względu na: cechą charakterystyczną, że te geny nie rekombinują i w rezultacie wykazują znacznie wyraźniejszą linię filogenetyczną niż wiele geny jądrowe. Efektywna wielkość populacji obliczona na podstawie genów mitochondrialnych (i funkcjonowania chromosomu Y w podobny sposób) wynosi około jednej czwartej wielkości obliczonej dla genów mitochondrialnych. genów jądrowych, wtedy dywergencja zachodzi prawie cztery razy szybciej niż w przypadku genów jądrowych, to szybkie tempo dywergencji (i przepływu genów) może spowodować obserwowany wzór dziedziczenia w tych jednorodzicielskich genach jest inny niż filogenezy uzyskane z genami jądrowymi (które stanowią większość puli genów w indywidualny).
Bibliografia
Achilli, A., Perego, U. A., Bravi, C. M., Coble, M. D., Kong, Q.-P. i in. (2008). Filogeneza czterech panamerykańskich haplogrup MtDNA: Implikacje dla badań nad ewolucją i chorobami. PLoS 1, 3(3), e1764.Cavalli-Sforza, L. L. (2000). Geny, narody i języki (1. edycja w Pocket Library wyd.). Barcelona, Hiszpania: krytyczny artykuł redakcyjny.
Rebato E., Susanne C. i Chiarelli B. (Edycje). (2005). Aby zrozumieć antropologię biologiczną: ewolucja i biologia człowieka. Słowo Boże.
Torroni, A., Schurr, T. G., Cabell, M. F, Brązowy, M. D, Neel, J. V., Larsen, M. i in. (1993). Powinowactwa azjatyckie i promieniowanie kontynentalne czterech założycieli rdzennych Amerykanów mtDNA. American Journal of Human Genetics, 53(3), 563.
(2022). mitochondrialny DNA, Narodowy Instytut Badań nad Genomem Człowieka.
-Schematyczne odniesienie do mitochondrialnego DNA-
Crespillo M., Paredes M., Arimany J., Guerrero L. i Valverde J. (2004). Hiszpańska wojna domowa (1936-1939): identyfikacja ludzkich szczątków z masowych grobów w Katalonii za pomocą analizy mitochondrialnego DNA. O sprawie. Zeszyty medycyny sądowej (38), 37-46.