Definição de DNA mitocondrial
Miscelânea / / July 04, 2022
definição de conceito
O genoma mitocondrial é uma pequena molécula circular de dupla hélice, fechada, de 16.569 pares de bases (pb) que é herdada apenas de mães para filhos e filhas (raramente). casos de heteroplasmia), portanto não há recombinação genética no momento da concepção, também evolui exclusivamente por acúmulo de mutações no tempo.






Lic. em Antropologia Física
A alta taxa de mutação do genoma mitocondrial (10 a 20 vezes maior do que o ADN nuclear, em termos de genes com funções comparáveis) é útil para diferenciar entre populações que ao longo do tempo foram biologicamente relacionados, no entanto, sua taxa de mutação pode ser tão rápida que fenômenos de mutação podem ocorrer. retromutação. A taxa de evolução A média desta molécula foi calculada a partir de espécies nas quais os tempos de divergência de restos fósseis estavam disponíveis, e de dados biodemográficos ou de proteínas; o resultado dá 1-2% por milhão de anos, válido para diferentes ordens.
Este DNA específico funciona como uma ferramenta para pesquisar parentesco biológico entre populações, pois suas propriedades permitem elucidar as relações entre populações. que divergiram nos últimos tempos, sem ter que considerar fenômenos de recombinação e adicionar uma dimensão temporário; Para isso, deve-se ter em mente o seguinte: dois indivíduos cujo ancestral comum foi uma mulher terão moléculas de DNA mitocondrial tão diferentes quanto o tempo passou desde a separação das antepassado.
Haplogrupos para classificação de DNA mitocondrial em distribuição geográfica
Algumas regiões do DNA podem ser muito semelhantes entre si, permitindo sua classificação no mesmo cluster, que é conhecido como um haplogrupo. Por exemplo, Torroni et al (1993) tipificaram as linhagens mitocondriais fundadoras para o continente americano, o que receberam a denominação de haplogrupos A, B, C e D, de acordo com as mutações pontuais da sequência mitocondrial. Essas mutações criam diferentes sítios de clivagem para enzimas específicas, conforme descrito a seguir: na linhagem A existe o sítio cutoff 663 para a enzima HaeIII, linhagem C é caracterizada pelo sítio 13259 para HincII, na linhagem D o sítio 5176 é reconhecido por AluI; esta identificação é feita a partir de polimorfismos de comprimento de fragmento (RFLP). No caso da linhagem B, há uma deleção de 9 pares de bases na posição 8272-8289.
A distribuição geográfica de cada linhagem foi assim descrita: a linhagem A é predominante no continente americano e especialmente na América do Norte; no entanto, este haplogrupo também é atribuído a populações mesoamericanas. As linhagens C e D aparecem principalmente na América do Sul; A linhagem B é encontrada na região norte e sul da costa do Pacífico, e Kemp et al (2010) a propõe como uma linhagem característica da família Yuto-asteca e sudoeste dos EUA
Além disso, existem mutações que são compartilhadas entre indivíduos de um mesmo haplogrupo, o que permitiu descrever haplótipos (ou sublinhagens) específicos de algumas populações. A Figura 1 mostra as regiões do DNA mitocondrial que contêm as mutações para reconhecer cada linhagem.
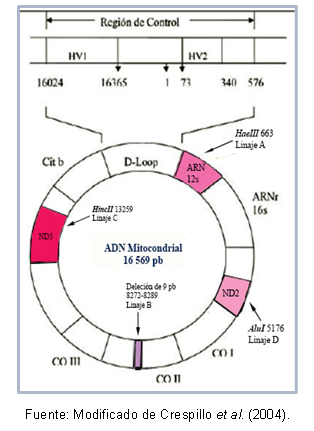
Figura 1. Regiões de DNA mitocondrial que tipificam cada linhagem fundadora americana.
Por alguns anos apenas essas quatro linhagens foram reconhecidas até que o haplogrupo X foi incluído para as populações do norte do continente; que também está distante das populações europeias. As linhagens anteriores têm correspondência com populações asiáticas, embora sejam observadas com menos frequência do que no continente americano; as linhagens A, B e C não são encontradas em africanos e caucasianos modernos; e a linhagem D também existe na África, mas associada a outros locais de restrição. Com o exposto, poderíamos dizer que esses haplogrupos caracterizam as populações americanas e, portanto, seu estudo é adequado em termos de povoamento e migrações (antigas e recentes).
distâncias genéticas
A maneira de estabelecer semelhanças ou diferenças genéticas entre populações é com distâncias genéticas, que podem ter um explicação histórica, uma vez que mudam (aumentam ou diminuem) ao longo das gerações, e podem nos levar a eventos na história de uma população por exemplo: no momento de uma grande migração ou contato entre duas culturas, com esse cálculo podemos discernir quais mecanismos entraram em ação para nos dar este ou aquele resultado.
Tanto a deriva genética quanto o fluxo gênico têm a ver com a facilidade com que um grupo, neste caso seres humanos, tem que se mover dentro de um grupo. território e entrar em contato com outros grupos. Assim, o isolamento geográfico é a distância genética que cresce com o aumento da distância geográfica entre os grupos humanos.
Hipótese da diversidade do DNA mitocondrial
Para explicar o diversidade nas linhagens mitocondriais fundadoras na América, existem dois hipótese: que este continente foi colonizado por múltiplos eventos da Beringia, ou que uma vez que a migração ocorreu, ocorreram mudanças evolutivas após a colonização. Também são explicadas duas vias de entrada dessas variações no continente, a primeira propõe que os quatro haplogrupos fundadores sem variações, ou seja, cada um com uma haplótipo radicular, poderiam ter chegado logo após o Último Máximo Glacial ou um pouco antes, com datas de 21 mil a 19 mil anos atrás e teriam seguido uma rota costeira pelo Pacífico; a segunda proposta sugere que essas variações intra-haplogrupo já existiam na Beringia e foram trazidas para o sul do continente americano, mas sua entrada teria sido exatamente no final do Último Máximo Glacial para que as rotas dentro do continente já estivessem livres, então a entrada desses grupos humanos seria 19 mil atrás anos. Há também uma grande diversidade de haplogrupo A e um tempo de coalescência para ele menor que os demais (17 mil anos).
A explicação mais provável para isso é que se deve a expansões secundárias do haplogrupo A da Beringia, muito depois do fim do Último Máximo Glacial. Apesar da discrepância quanto à época de entrada do homem nas Américas, estudos genéticos têm dão alguma clareza, pois sustentam a hipótese de que havia grupos humanos no continente americano antes Clóvis; e verifica-se que há uma separação entre os ancestrais do nordeste da Ásia há 25-35 mil anos e a entrada na América há 15-35 mil anos. A Figura 2 mostra as rotas dos haplogrupos mitocondriais no mundo e o tempo de divergência em anos anteriores ao presente.
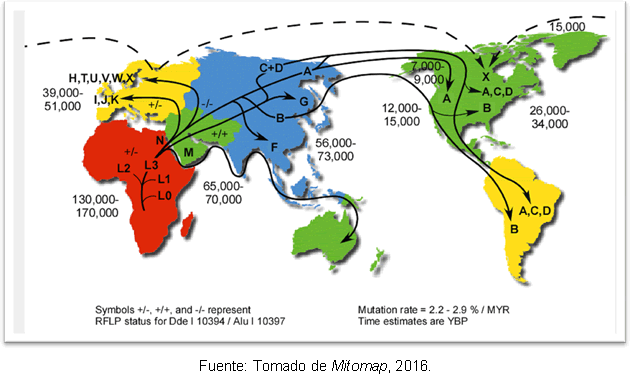
Figura 2. Mapa das diferentes rotas de dispersão de linhagens mitocondriais
Importância da filogeografia no estudo genético e dados de genes mitocondriais
Existe uma ferramenta muito útil para análise genética; a filogeografia. Estas são as primeiras aplicações de estudos moleculares, e com isso buscamos poder determinar as relações filogenéticas e espaciais entre sequências de nucleotídeos, neste caso o DNA mitocondrial. A distribuição espacial pode se assemelhar a um padrão temporal, ou seja, as sequências de DNA geograficamente mais distantes devem ser as mais geneticamente diferentes, bem como sequências de DNA que divergiram há muito tempo também devem ser as mais diferentes geneticamente. Assim, populações geograficamente distantes, com pouco ou nenhum fluxo gênico entre elas, acumulariam diferenças devido à deriva e mutação genética, mesmo por seleção; mas podem ocorrer situações que não permitem a deriva, como um ou mais efeitos fundadores ou outros padrões de fluxo gênico.
Os dados fornecidos pelos genes mitocondriais estão entre os mais úteis na pesquisa de filogeografia devido à característica que esses genes têm de não se recombinarem e como resultado eles mostram uma linha filogenética muito mais clara do que muitos genes nucleares. O tamanho efetivo da população calculado com os genes mitocondriais (e com o cromossomo Y funcionando de maneira semelhante) é cerca de um quarto do calculado para os genes mitocondriais. genes nucleares, então a divergência ocorre quase quatro vezes mais rápido do que com genes nucleares, essa rápida taxa de divergência (e fluxo gênico) pode causar a O padrão de herança observado nesses genes uniparentais é diferente das filogenias obtidas com genes nucleares (que representam a maioria do pool gênico em uma Individual).
Referências
Achilli, A., Perego, U. A., Bravi, C. M., Coble, M. D., Kong, Q.-P., & et ai. (2008). A Filogenia dos Quatro Haplogrupos Pan-Americanos do MtDNA: Implicações para Estudos Evolutivos e de Doenças. PLoS ONE, 3(3), e1764.Cavalli-Sforza, L. EU. (2000). Genes, povos e línguas (1ª Edição em Pocket Library ed.). Barcelona, Espanha: Editorial Crítica.
Rebato, E., Susanne, C., & Chiarelli, B. (Editar% s.). (2005). Compreender a Antropologia Biológica: evolução e biologia humana. Palavra Divina.
Torroni, A., Schurr, T. G., Cabell, M. F, Brown, M. D, Neel, J. V., Larsen, M., et ai. (1993). Afinidades asiáticas e radiação continental dos quatro mtDNAs nativos americanos fundadores. The American Journal of Human Genetics, 53(3), 563.
(2022). DNA mitocondrial, Instituto Nacional de Pesquisa do Genoma Humano.
-Referência esquemática do DNA mitocondrial-
Crespillo, M., Paredes, M., Arimany, J., Guerrero, L., & Valverde, J. (2004). Guerra Civil Espanhola (1936-1939): identificação de restos humanos de valas comuns na Catalunha por análise de DNA mitocondrial. Sobre um caso. Cadernos de Medicina Legal (38), 37-46.



