Definisjon av mitokondrielt DNA
Miscellanea / / July 04, 2022
begrepsdefinisjon
Det mitokondrielle genomet er et lite, lukket sirkulært molekyl med dobbel helix på 16 569 basepar (bp) som bare arves fra mødre til sønner og døtre (sjelden). tilfeller av heteroplasmi), så det er ingen genetisk rekombinasjon på unnfangelsestidspunktet, den utvikler seg også utelukkende ved akkumulering av mutasjoner i vær.






Lic. i fysisk antropologi
Den høye mutasjonshastigheten til mitokondrie-genomet (10 til 20 ganger den av DNA kjernefysisk, når det gjelder gener med sammenlignbare funksjoner) er nyttig for å skille mellom populasjoner som over tid har vært biologisk relatert, men mutasjonshastigheten deres kan være så høy at mutasjonsfenomener kan oppstå. tilbakemutasjon. Satsen på utvikling Gjennomsnittet av dette molekylet er beregnet fra arter der avvikstider fra fossile rester var tilgjengelige, og fra biodemografiske eller proteindata; resultatet gir 1-2 % per million år, gyldig for ulike ordrer.
Denne spesielle DNA fungerer som et verktøy for å
undersøkelser det biologiske slektskapet mellom populasjoner fordi deres egenskaper gjør det mulig å belyse forholdet mellom populasjoner som har divergert i nyere tid, uten å måtte vurdere rekombinasjonsfenomener og legge til en dimensjon midlertidig; For dette må følgende huskes: to individer hvis felles stamfar har vært en kvinne vil ha mitokondrielle DNA-molekyler så forskjellige som tiden har gått siden separasjonen av stamfar.Haplogrupper for mitokondriell DNA-klassifisering i geografisk distribusjon
Noen regioner av DNA kan være svært like hverandre, noe som tillater deres klassifisering i samme klynge, som er kjent som en haplogruppe. For eksempel karakteriserte Torroni et al (1993) de grunnleggende mitokondrielle linjene for det amerikanske kontinentet, som mottok betegnelsen haplogruppe A, B, C og D, i henhold til punktmutasjoner av sekvensen mitokondrie. Disse mutasjonene skaper forskjellige spaltningssteder for spesifikke enzymer, som beskrevet nedenfor: i avstamning A er stedet cutoff 663 for enzymet HaeIII, avstamning C er karakterisert ved sted 13259 for HincII, i avstamning D er sted 5176 gjenkjent for AluI; denne identifiseringen er laget fra fragmentlengde polymorfismer (RFLP). Når det gjelder avstamning B, er det en sletting av 9 basepar ved posisjon 8272-8289.
Den geografiske fordelingen av hver avstamning har blitt beskrevet som følger: avstamning A er dominerende på det amerikanske kontinentet og spesielt i Nord-Amerika; Imidlertid tilskrives denne haplogruppen også mesoamerikanske populasjoner. Linjene C og D vises hovedsakelig i Sør-Amerika; avstamning B finnes i den nordlige og sørlige regionen av Stillehavskysten, og Kemp et al (2010) foreslår den som en karakteristisk avstamning av familie Yuto-aztekanske og sørvestlige U.S.A.
I tillegg er det mutasjoner som deles mellom individer av samme haplogruppe, noe som har gjort det mulig å beskrive spesifikke haplotyper (eller sublineages) av enkelte populasjoner. Figur 1 viser områdene i mitokondrielt DNA som inneholder mutasjonene for å gjenkjenne hver avstamning.
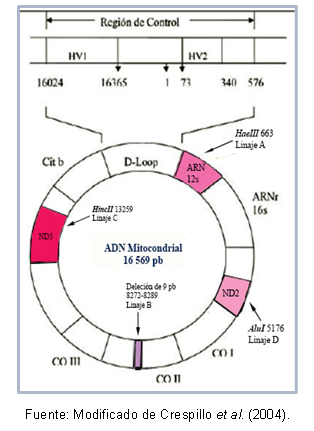
Figur 1. Mitokondrielle DNA-regioner som kjennetegner hver amerikansk grunnleggende avstamning.
I noen år ble bare disse fire linjene anerkjent inntil haplogruppe X ble inkludert for de nordlige populasjonene på kontinentet; som også er fjernt beslektet med europeiske befolkninger. De tidligere slektene har en korrespondanse med asiatiske populasjoner, selv om de er observert sjeldnere enn på det amerikanske kontinentet; slekt A, B og C finnes ikke hos moderne afrikanere og kaukasiere; og avstamning D eksisterer også i Afrika, men assosiert med andre restriksjonssteder. Med ovenstående kan vi si at disse haplogruppene kjennetegner de amerikanske populasjonene og derfor er studien deres tilstrekkelig med tanke på bosetting og migrasjoner (gamle og nyere).
genetiske avstander
Måten å etablere genetiske likheter eller forskjeller mellom populasjoner er med genetiske avstander, som kan ha en historisk forklaring, siden de endres (øker eller reduseres) over generasjonene, og kan føre oss til hendelser i historien av en befolkning for eksempel: på tidspunktet for en stor migrasjon eller kontakt mellom to kulturer, med denne beregningen kan vi skjelne hvilke mekanismer som kom inn for å gi oss dette eller det resultatet.
Både genetisk drift og genflyt har å gjøre med hvor lett en gruppe, i dette tilfellet mennesker, må bevege seg innenfor en gruppe. territorium og komme i kontakt med andre grupper. Så, geografisk isolasjon er den genetiske avstanden som vokser med økende geografisk avstand mellom menneskelige grupper.
Mitokondriell DNA-diversitetshypotese
For å forklare mangfold i de grunnleggende mitokondrielinjene i Amerika er det to hypotese: at dette kontinentet har blitt kolonisert av flere hendelser fra Beringia, eller at når migrasjon har skjedd, har evolusjonære endringer skjedd etter koloniseringen. To inngangsveier for disse variasjonene til kontinentet er også forklart, den første foreslår at de fire grunnleggende haplogruppene uten variasjoner, det vil si hver med en rothaplotype, kunne de ha kommet like etter det siste istidsmaksimum eller litt før, med datoer for 21 tusen til 19 tusen år siden og ville ha fulgt en kystrute ved Fredelig; det andre forslaget antyder at disse intra-haplogruppe-variasjonene allerede eksisterte i Beringia og ble brakt til sør på det amerikanske kontinentet, men deres inntreden ville ha vært nøyaktig på slutten av det siste istidsmaksimum, slik at rutene innenfor kontinentet allerede ville være frie, så ville inngangen til disse menneskegruppene være for 19 tusen siden år. Det er også et stort mangfold av haplogruppe A og en koalescenstid for den som er kortere enn resten (17 tusen år).
Den mest sannsynlige forklaringen på dette er at det skyldes sekundære utvidelser av haplogruppe A fra Beringia, lenge etter slutten av det siste istidsmaksimum. Til tross for uoverensstemmelsen angående tidspunktet for menneskets inntog i Amerika, har genetiske studier gjort det gi litt klarhet, fordi de støtter hypotesen om at det fantes menneskegrupper på det amerikanske kontinentet før Clovis; og det er funnet at det er et skille mellom forfedrene til Nordøst-Asia for 25-35 tusen år siden og inntoget i Amerika for 15-35 tusen år siden. Figur 2 viser rutene til de mitokondrielle haplogruppene i verden og tidspunktet for divergens i år før nåtid.
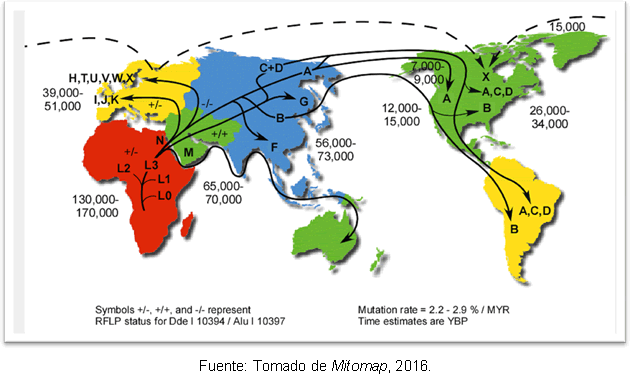
Figur 2. Kart over de forskjellige spredningsrutene for mitokondrielle avstamninger
Betydningen av fylogeografi i genetiske studier og mitokondrielle gendata
Det finnes et veldig nyttig verktøy for genetisk analyse; fylogeneografien. Dette er de første anvendelsene av molekylære studier, og med det søker vi å kunne bestemme de fylogenetiske og romlige forholdene mellom nukleotidsekvenser, i dette tilfellet DNA mitokondrie. Den romlige fordelingen kan ligne et tidsmønster, det vil si at de mest geografisk fjerne DNA-sekvensene bør være mest genetisk forskjellige, samt DNA-sekvenser som divergerte for lenge siden burde også være de mest forskjellige genetisk. Så geografisk fjerne populasjoner, med liten eller ingen genstrøm mellom dem, ville akkumulere forskjeller på grunn av genetisk drift og mutasjon, selv ved seleksjon; men det kan oppstå situasjoner som ikke tillater drift, for eksempel en eller flere grunnleggereffekter eller andre mønstre av genstrøm.
Dataene levert av mitokondrielle gener har vært blant de mest nyttige i fylogeografiforskning på grunn av karakteristisk at disse genene ikke rekombinerer og som et resultat viser de en mye klarere fylogenetisk linje enn mange kjernefysiske gener. Den effektive populasjonsstørrelsen beregnet med mitokondrie-genene (og med Y-kromosomet som fungerer på lignende måte) er omtrent en fjerdedel av den som er beregnet for mitokondrie-genene. kjernefysiske gener, da divergens skjer nesten fire ganger raskere enn med kjernefysiske gener, kan denne raske divergenshastigheten (og genstrømmen) forårsake observert arvemønster i disse uniparentale genene er annerledes enn fylogeniene oppnådd med kjernefysiske gener (som representerer størstedelen av genmassen i en individuell).
Referanser
Achilli, A., Perego, U. A., Bravi, C. M., Coble, M. D., Kong, Q.-P., & et al. (2008). Fylogenien til de fire panamerikanske MtDNA-haplogruppene: Implikasjoner for evolusjons- og sykdomsstudier. PLoS ONE, 3(3), e1764.Cavalli-Sforza, L. L. (2000). Gener, folkeslag og språk (1st Edition i Pocket Library ed.). Barcelona, Spania: Kritisk redaksjon.
Rebato, E., Susanne, C., & Chiarelli, B. (Redigeringer.). (2005). For å forstå biologisk antropologi: evolusjon og menneskelig biologi. Guddommelige Ord.
Torroni, A., Schurr, T. G., Cabell, M. F, Brown, M. D, Neel, J. V., Larsen, M., et al. (1993). Asiatiske affiniteter og kontinental stråling av de fire grunnleggende indianske mtDNA-ene. The American Journal of Human Genetics, 53(3), 563.
(2022). mitokondrielt DNA, National Human Genome Research Institute.
-Mitokondriell DNA skjematisk referanse-
Crespillo, M., Paredes, M., Arimany, J., Guerrero, L., & Valverde, J. (2004). Spansk borgerkrig (1936-1939): identifikasjon av menneskelige levninger fra massegraver i Catalonia ved mitokondriell DNA-analyse. Om en sak. Rettsmedisinske notatbøker (38), 37-46.


